Das als COVID-19 bekannte Schwere Akute Atemwegssyndrom – verursacht durch das SARS-CoV-2-Virus – verbreitet sich nachweislich über Tröpfcheninfektion und engen Kontakt.[1] Die Belastung durch COVID-19 war in der Lombardei und der Poebene (Norditalien)[2] besonders hoch, einem Gebiet mit hohen Feinstaubkonzentrationen, von denen bereits bekannt ist, dass sie negative Auswirkungen auf die menschliche Gesundheit haben.[3] Regionale Zahlen für Italien vom 12. April zeigen, dass etwa 30 % der derzeit positiv getesteten Personen noch immer in der Lombardei leben (etwa 40 %, wenn man die Gesamtzahl der seit Beginn der Epidemie bestätigten Fälle berücksichtigt), gefolgt von der Emilia-Romagna (13,5 %), Piemont (10,5 %) und Venetien (10 %).[2] Auf diese vier Regionen der Poebene entfallen 80 % aller in Italien registrierten Todesfälle und 65 % der Einweisungen auf Intensivstationen.[2]
Eine von der Harvard School of Public Health durchgeführte Studie scheint einen Zusammenhang zwischen dem Anstieg der PM-Konzentrationen und der Sterblichkeitsrate aufgrund von COVID-19 in den USA zu bestätigen[4]. In früheren Mitteilungen haben wir die Möglichkeit in Betracht gezogen, dass das SARS-CoV-2-Virus während der Ausbreitung der Infektion auf Feinstaub (PM) vorhanden sein könnte[5,6], was im Einklang mit bereits vorliegenden Beweisen steht
für andere Viren verfügbar.[7-15] Allerdings ist die Frage des mit Feinstaub in der Luft verbundenen Mikrobioms, insbesondere in städtischen Umgebungen, noch weitgehend unerforscht,[16] und bis heute hat noch niemand experimentelle Studien durchgeführt, die speziell darauf abzielen, das Vorhandensein von SARS-CoV-2 auf Feinstaub zu bestätigen oder auszuschließen.
Hier präsentieren wir die ersten Ergebnisse der Analysen, die wir an 34 PM10-Proben von PM10 im Freien/in der Luft von einem Industriegelände in der Provinz Bergamo durchgeführt haben. Die Proben wurden mit zwei verschiedenen Luftprobenehmern über einen Zeitraum von drei Wochen vom 21. Februar bis 13. März gesammelt.
Nach der von Pan et al. im Jahr 2019 beschriebenen Methodik (zur Sammlung, Partikelgrößenbestimmung und Erkennung luftgetragener Viren)[17] wurden PM-Proben auf Quarzfaserfiltern mithilfe eines gravimetrischen Luftprobenehmers mit geringem Volumen (38,3 l/min für 23 h) gesammelt, der der Referenzmethode EN12341:2014 zur PM10-Überwachung entspricht. Partikel wurden auf Filtern mit 99,9 % typischerAerosolretention, ordnungsgemäß gelagert und an das Labor für Angewandte und Vergleichende Genomik der Universität Triest geliefert. Da die Probe vermutlich reich an DNA-Polymerase-Inhibitoren war, führten wir die RNA-Extraktion mithilfe des Quick-RNA-Kits für Stuhlmikroben durch, das an den Filtertyp angepasst war.[18] Ein halber Filter wurde mit der Oberseite nach innen gerollt.in einem 5-ml-Polypropylenröhrchen zusammen mit den im Kit enthaltenen Beads. Aus dem anfänglichen 1 ml Lysepuffer konnten wir etwa 400 µl Lösung gewinnen, die anschließend gemäß den Standardprotokollen verarbeitet wurden, was zu einem finalen Eluat von 15 µl führte. Anschließend wurden 5 µl für den SARS-CoV-2-Test verwendet. Aufgrund der besonderen Herkunft der Probe wurde der qScript XLT 1-Step RT-qPCR ToughMix verwendet. [19] Die Amplifikationssysteme entsprachen denen des von Corman et al. entwickelten und auf der WHO-Website veröffentlichten Protokolls [20].
Der Test zielte explizit darauf ab, das Vorhandensein von SARS-CoV-2-RNA auf Feinstaub zu bestätigen oder auszuschließen. Die erste Analyse nutzte das „E-Gen“ als molekularen Marker und lieferte auf 15 von 16 Filtern ein beeindruckendes positives Ergebnis, auch wenn der Ct-Wert erwartungsgemäß zwischen 36 und 38 Zyklen lag.
Anschließend haben wir die Analyse an 6 der positiven Filter (die bereits positiv auf das „E-Gen“ reagierten) wiederholt, indem wir das „RtDR-Gen“ als molekularen Marker verwendeten – das hochspezifisch für SARS-CoV-2 ist – und dabei 5 signifikante Positivergebnisse erzielten. Kontrolltests zum Ausschluss falsch positiver Ergebnisse wurden ebenfalls erfolgreich durchgeführt (Abb. 1).
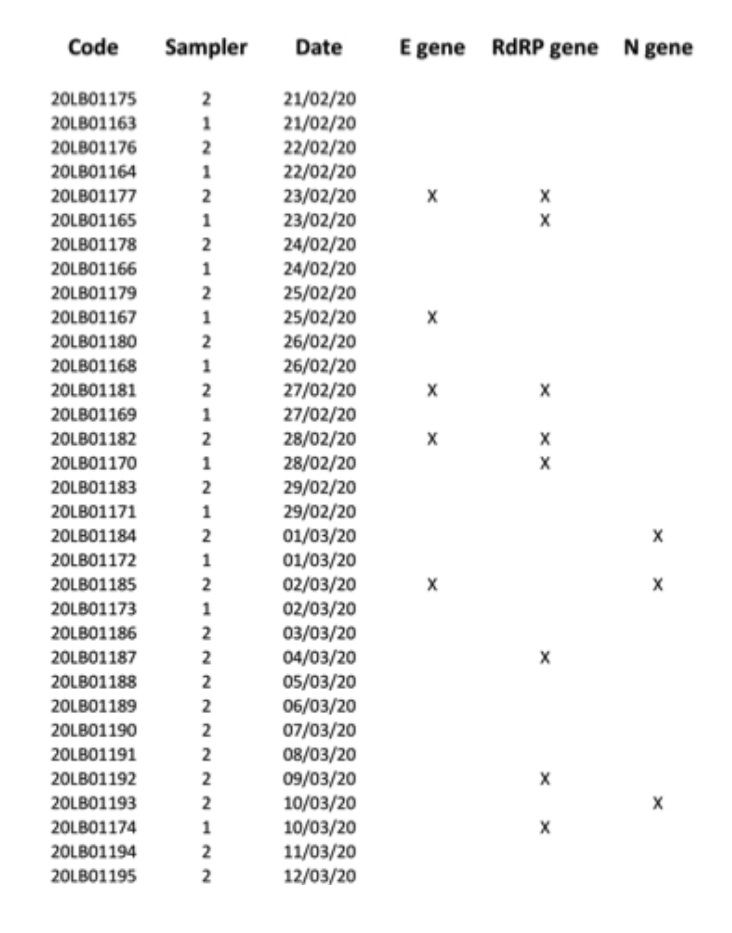
Um das knappe Probenmaterial nicht zu verknappen, wurden die restlichen extrahierten RNAs an das örtliche Universitätsklinikum (eines der von der italienischen Regierung für SARS-CoV-2-Diagnosetests zugelassenen klinischen Zentren) geliefert, um einen zweiten parallelen Blindtest durchzuführen. Dieses zweite klinische Labor testete 34 RNA-Extraktionen auf die Gene E, N und RdRP und meldete sieben positive Ergebnisse für mindestens eines der drei Markergene, wobei die Positivität für alle drei Marker separat bestätigt wurde (Abb. 2). Aufgrund der Art der Probe und in Anbetracht der Tatsache, dass die Probenahme nicht für klinisch-diagnostische Zwecke, sondern für Umweltverschmutzungstests durchgeführt wurde (unter Berücksichtigung der Tatsache, dass die Filter mindestens vier Wochen lang gelagert wurden, bevor sie molekulargenetischen Analysen unterzogen wurden, wieAufgrund des Lockdowns in Italien konnten wir bestätigen, dass wir das Vorhandensein von SARS-CoV-2-Virus-RNA durch den Nachweis des hochspezifischen „RtDR-Gens“ auf acht Filtern nachgewiesen haben. Da jedoch zusätzliches Filtermaterial fehlte, konnten wir nicht genügend Tests wiederholen, um für alle drei molekularen Marker gleichzeitig ein positives Ergebnis zu erzielen.
Dies ist der erste vorläufige Beweis dafür, dass SARS-CoV-2-RNA auf Feinstaub im Freien vorhanden sein kann. Dies deutet darauf hin, dass SARS-CoV-2 unter Bedingungen atmosphärischer Stabilität und hoher Feinstaubkonzentrationen Cluster mit Feinstaub im Freien bilden und – durch Verringerung ihres Diffusionskoeffizienten – die Persistenz des Virus in der Atmosphäre erhöhen könnte. Weitere Bestätigungen dieser vorläufigenDie Untersuchungen laufen und sollten eine Echtzeitbewertung der Vitalität von SARS-CoV-2 sowie seiner Virulenz bei Adsorption an Feinstaub umfassen. Derzeit können keine Annahmen über den Zusammenhang zwischen dem Vorhandensein des Virus auf Feinstaub und dem Verlauf des COVID-19-Ausbruchs getroffen werden. Weitere Fragen, die speziell behandelt werden müssen, sind die durchschnittlichen Feinstaubkonzentrationen, die letztendlichfür einen möglichen „Boost-Effekt“ der Ansteckung erforderlich (falls bestätigt wird, dass PM als „Träger“ für die Kerne der viralen Tröpfchen fungieren könnte) oder sogar die theoretische Möglichkeit einer Immunisierung infolge minimaler Dosen bei niedrigeren PM-Schwellenwerten.
Abb.1 Amplifikationskurven der E- (A) und RdRP-Gene (B): grüne Linien stellen getestete Filter dar; Querlinienstellt Referenzfilterextraktionen dar; rote Linien stellen die Amplifikation der positiven Proben dar.
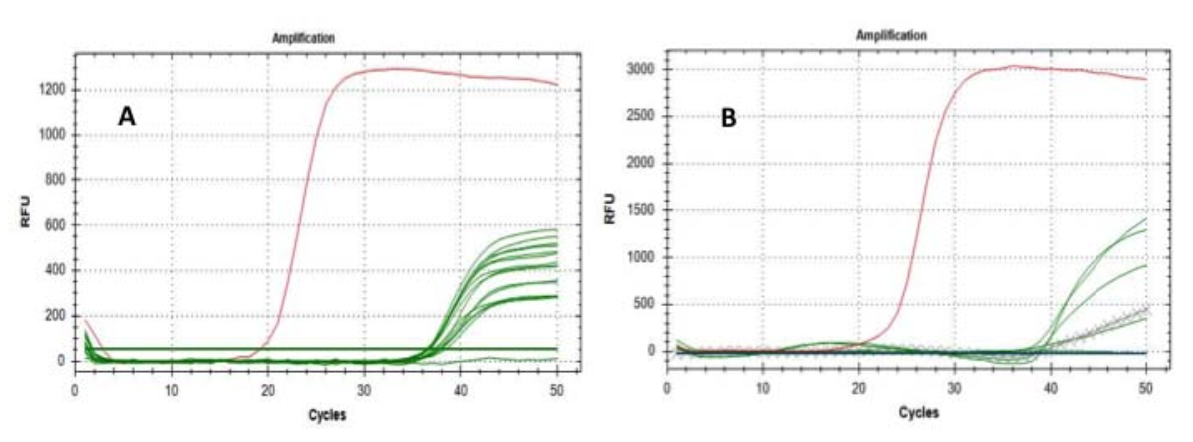
Abb. 2. Positive Ergebnisse (markiert mit X) für E-, N- und RdRP-Gene, die für alle 34 PM10-Proben erhalten wurdenFilter, die in den zweiten parallelen Analysen getestet wurden.
 Leonardo Setti1, Fabrizio Passarini2, Gianluigi De Gennaro3, Pierluigi Barbieri4, Maria Grazia Perrone5, Massimo Borelli6, Jolanda Palmisani3, Alessia Di Gilio3, Valentina Torboli6, Alberto Pallavicini6, Maurizio Ruscio7, Prisco Piscitelli8, Alessandro Miani8,9
Leonardo Setti1, Fabrizio Passarini2, Gianluigi De Gennaro3, Pierluigi Barbieri4, Maria Grazia Perrone5, Massimo Borelli6, Jolanda Palmisani3, Alessia Di Gilio3, Valentina Torboli6, Alberto Pallavicini6, Maurizio Ruscio7, Prisco Piscitelli8, Alessandro Miani8,9
1. Abteilung für Industrielle Chemie, Universität Bologna, Viale del Risorgimento – 4, I-40136, Bologna, Italien
e-mail: leonardo.setti@unibo.it
2. Interdepartementales Zentrum für Industrieforschung „Erneuerbare Energien, Umwelt, Blaues Wachstum, Energie“,
University of Bologna, Rimini, Italy e-mail: fabrizio.passarini@unibo.it
3. Fakultät für Biologie, Universität „Aldo Moro“ Bari, Bari, Italien
e-mail: gianluigi.degennaro@uniba.it; alessia.digilio@uniba.it; jolanda.palmisani@uniba.it
4. Fakultät für Chemische und Pharmazeutische Wissenschaften, Universität Triest, Triest, Italien
e-mail: barbierp@units.it
5. Abteilung für Umweltforschung, TCR TECORA, Mailand, Italien
e-mail: mariagrazia.perrone@tcrtecora.com
6. Fakultät für Biowissenschaften – Universität Triest, Triest, Italien
e-mail: borelli@units.it; torboli@units.it; pallavic@units.it
7. Abteilung für Labormedizin, Universitätsklinikum Giuliano Isontina (ASU GI), Triest, Italien
email: maurizio.ruscio@asugi.sanita.fvg.it
8. Italienische Gesellschaft für Umweltmedizin (SIMA), Mailand, Italien
e-mail: priscofreedom@hotmail.com; alessandro.miani@unimi.it
9. Institut für Umweltwissenschaften und -politik, Universität Mailand, Mailand, Italien
e-mail: priscofreedom@hotmail.com; alessandro.miani@unimi.it
Korrespondierender Autor:
Leonardo Setti, Department of Industrial Chemistry, University of Bologna Viale del Risorgimento 4, 40136, Bologna, Italy; e-mail: leonardo.setti@unibo.it
Verweise
1. Weltgesundheitsorganisation, Übertragungswege des Virus, das COVID-19 verursacht: Auswirkungen auf die Vorsorgeempfehlungen des IPC, Wissenschaftliche Kurzfassung; verfügbar unter: https://www.who.int/newsroom/commentaries/detail/modes-of-transmission-of-virus-causing-covid-19-implications-for-ipcprecaution-recommendations (29. März 2020)
2. Italienisches Gesundheitsministerium, tägliches Bulletin zum Covid-19-Ausbruch in Italien, verfügbar unter http://www.salute.gov.it/imgs/C_17_notizie_4451_0_file.pdf
3. EEA, Europäische Umweltagentur, Bericht zur Luftqualität in Europa 2019; Nr. 10/2019; Europäische Umweltagentur: Kopenhagen, Dänemark, verfügbar unter: https://www.eea.europa.eu/publications/airquality-in-europe-2019
4. Xiao Wu, Rachel C. Nethery, M. Benjamin Sabath, Danielle Braun, Francesca Dominici, Exposition gegenüber Luftverschmutzung und COVID-19-Sterblichkeit in den Vereinigten Staaten, verfügbar unter: https://projects.iq.harvard.edu/files/covid-pm/files/pm_and_covid_mortality.pdf
5. Italienische Gesellschaft für Umweltmedizin (SIMA), Positionspapier Feinstaub und COVID-19,
verfügbar unter: http://www.simaonlus.it/wpsima/wp-content/uploads/2020/03/COVID_19_positionpaper_ENG.pdf
6. Setti L., Passarini F., De Gennaro G., Barbieri P., Perrone MG, Piazzalunga A., Borelli M., Palmisani J., Di Gilio A, Piscitelli P, Miani A., Is there a Plausible Role for Particulate Matter in the spreading of COVID-19 in Northern Italy?, BMJ Rapid Responses, 8. April 2020, verfügbar unter: https://www.bmj.com/content/368/bmj.m1103/rapid-responses
7. Sedlmaier, N., Hoppenheidt, K., Krist, H., Lehmann, S., Lang, H., Buttner, M. Entstehung von mit dem Vogelgrippevirus (AIV) kontaminiertem fäkalem Feinstaub (PM2,5): Genom- und Infektiositätsnachweis sowie Berechnung der Immission. Veterinärmikrobiologie. 139, 156-164 (2009)
8. Zhao, Y., Richardson, B., Takle, E., Chai, L., Schmitt, D., Win, H. Die Übertragung über die Luft könnte bei der Ausbreitung der hochpathogenen Vogelgrippe-Ausbrüche in den Vereinigten Staaten im Jahr 2015 eine Rolle gespielt haben. Sci Rep. 9, 11755. https://doi.org/10.1038/s41598-019-47788-z (2019)
9. Ma, Y., Zhou, J., Yang, S., Zhao, Y., Zheng, X. Bewertung der Auswirkungen von Staubereignissen auf die Maserninzidenz in Westchina. Atmosphärische Umwelt. 157, 1-9 (2017)
10. Sorensen, JH, Mackay, DKJ, Jensen, C. Ø., Donaldson, AI Ein integriertes Modell zur Vorhersage der atmosphärischen Ausbreitung des Maul- und Klauenseuchevirus Epidemiol. Infect., 124, 577–590 (2000)
11. Glostera, J., Alexandersen, S. Neue Richtungen: Luftgetragene Übertragung des Maul- und Klauenseuchevirus in der Atmosphäre, 38 (3), 503-505 (2004)
12. Reche, I., D'Orta, G., Mladenov, N., Winget, DM, Suttle, CA Ablagerungsraten von Viren und Bakterien oberhalb der atmosphärischen Grenzschicht. The ISME Journal. 12, 1154-1162 (2018)
13. Qin, N., Liang, P., Wu, C., Wang, G., Xu, Q., Xiong, X., Wang, T., Zolfo, M., Segata, N., Qin, H., Knight, R., Gilbert, JA, Zhu, TF Längsschnittuntersuchung des Mikrobioms im Zusammenhang mit Feinstaub in einer Megastadt. Genome Biology. 21, 55 (2020)
14. Zhao, Y., Richardson, B., Takle, E., Chai, L., Schmitt, D., Win, H. Die Übertragung über die Luft kann
spielte eine Rolle bei der Ausbreitung der hochpathogenen Vogelgrippe im Jahr 2015 in den Vereinigten Staaten. Sci
Rep. 9, 11755. https://doi.org/10.1038/s41598-019-47788-z (2019)
15. Ma, Y., Zhou, J., Yang, S., Zhao, Y., Zheng, X. Bewertung der Auswirkungen von Staubereignissen auf die Maserninzidenz in Westchina. Atmosphärische Umwelt. 157, 1-9 (2017)
16. Jiang, W., Laing, P., Wang, B., Fang, J.,Lang, J., Tian, G., Jiang, J., Zhu, TF Optimierte DNA-Extraktion und metagenomische Sequenzierung von luftgetragenen mikrobiellen Gemeinschaften. Nat. Protoc. 10, 768-779 (2015)
17. Pan, M., Lednicky, JA, Wu, C.-Y., Erfassung, Partikelgrößenbestimmung und Nachweis luftgetragener Viren. Journal of Applied Microbiology, 127, 1596-1611 (2019)
18. Zymoresearch Ldt, Produktbeschreibung, verfügbar unter: https://www.zymoresearch.com/products/quick-rnafecal-soil-microbe microprep-kit
19. Quantabio Ltd, Produktbeschreibung, verfügbar unter: https://www.quantabio.com/qscript-xlt-1-steprt-qpcr-toughmix
20. Corman, VM, Landt, O., Kaiser, M., Molenkamp, R., Meijer, A., Chu, DK und Mulders, DG (2020).
Nachweis des neuartigen Coronavirus 2019 (2019-nCoV) mittels Echtzeit-RT-PCR. Eurosurveillance, 25(3), verfügbar unter: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6988269/
Original: https://doi.org/10.1101/2020.04.15.20065995
Veröffentlichungszeit: 18. April 2020








