Ciężki ostry zespół oddechowy znany jako choroba COVID-19 – wywołany wirusem SARS-CoV-2 – rozprzestrzenia się drogą kropelkową i przez bliskie kontakty.[1] Obciążenie COVID-19 było wyjątkowo poważne w Lombardii i Dolinie Padu (północne Włochy),[2] obszarze charakteryzującym się wysokim stężeniem cząstek stałych, o których wiadomo, że mają negatywny wpływ na zdrowie ludzi.[3] Dane regionalne dostępne dla Włoch na dzień 12 kwietnia pokazują, że około 30% obecnie pozytywnych osób nadal mieszka w Lombardii (około 40%, jeśli wziąć pod uwagę wszystkie przypadki potwierdzone od początku epidemii), a następnie w Emilii-Romanii (13,5%), Piemoncie (10,5%) i Wenecji Euganejskiej (10%).[2] Na te cztery regiony Doliny Padu przypada 80% wszystkich zgonów odnotowanych we Włoszech i 65% przyjęć na oddziały intensywnej terapii.[2]
Badania przeprowadzone przez Harvard School of Public Health zdają się potwierdzać związek między wzrostem stężeń PM a wskaźnikami śmiertelności z powodu COVID-19 w USA[4]. W poprzednich komunikatach wysunęliśmy hipotezę, że wirus SARS-CoV-2 może być obecny na cząstkach stałych (PM) podczas rozprzestrzeniania się infekcji[5,6], co jest zgodne z dowodami już przedstawionymi
dostępne dla innych wirusów.[7-15] Jednak kwestia mikrobiomu związanego z pyłami zawieszonymi w powietrzu, zwłaszcza w środowiskach miejskich, pozostaje w dużej mierze niedostatecznie zbadana[16] i – jak dotąd – nikt nie przeprowadził badań eksperymentalnych mających na celu potwierdzenie lub wykluczenie obecności SARS-CoV-2 na pyłach zawieszonych.
W niniejszym artykule przedstawiamy pierwsze wyniki analiz, które przeprowadziliśmy na 34 próbkach pyłu PM10 unoszącego się w powietrzu i na zewnątrz obiektu przemysłowego w prowincji Bergamo, pobranych za pomocą dwóch różnych próbników powietrza w okresie 3 tygodni od 21 lutego do 13 marca.
Zgodnie z metodologią opisaną przez Pan i in. w 2019 r. (dotyczącą zbierania, określania wielkości cząstek i wykrywania wirusów przenoszonych drogą powietrzną)[17] próbki PM pobrano na filtrach z włókien kwarcowych przy użyciu niskoobjętościowego próbnika powietrza grawimetrycznego (38,3 l/min przez 23 godz.), zgodnego z metodą referencyjną EN12341:2014 do monitorowania PM10. Cząstki stałe zostały zatrzymane na filtrach z 99,9% typowychretencja aerozolu, odpowiednio przechowywana i dostarczona do laboratorium genomiki stosowanej i porównawczej Uniwersytetu w Trieście. Biorąc pod uwagę „środowiskowy” charakter próbki, prawdopodobnie bogatej w inhibitory polimeraz DNA, przystąpiliśmy do ekstrakcji RNA przy użyciu zestawu Quick RNA fecal soil microbe kit dostosowanego do rodzaju filtrów.[18] Połowa filtra została zwinięta, górną stroną skierowaną do wewnątrz,w 5 ml probówce polipropylenowej, wraz z kulkami dostarczonymi w zestawie. Z początkowego 1 ml buforu lizy udało nam się uzyskać około 400 ul roztworu, który następnie przetworzono zgodnie ze standardowymi protokołami, co dało końcowy eluat 15 ul. Następnie 5 ul użyto do testu SARS-CoV-2. Biorąc pod uwagę szczególne pochodzenie próbki, użyto qScript XLT 1-Step RT-qPCR ToughMix.[19] Systemy amplifikacji były systemami protokołu opracowanego przez Cormana i in., opublikowanymi na stronie internetowej WHO [20].
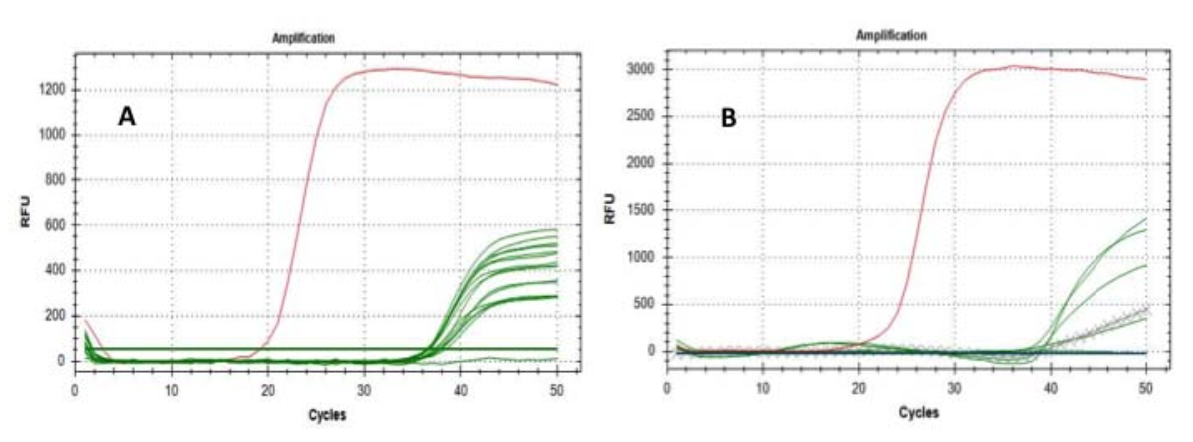
Test miał wyraźnie na celu potwierdzenie lub wykluczenie obecności RNA SARS-CoV-2 na cząstkach stałych. Pierwsza analiza wykorzystała „gen E” jako marker molekularny i dała imponujący wynik pozytywny na 15 z 16 filtrów, nawet jeśli, jak można było się spodziewać, Ct wynosiło od 36 do 38 cykli.
Następnie powtórzyliśmy analizę na 6 pozytywnych filtrach (już pozytywnych na „gen E”), używając „genu RtDR” jako markera molekularnego – który jest wysoce specyficzny dla SARS-CoV-2 – uzyskując 5 istotnych wyników pozytywnych; pomyślnie przeprowadzono również testy kontrolne w celu wykluczenia fałszywie pozytywnych wyników (ryc. 1).
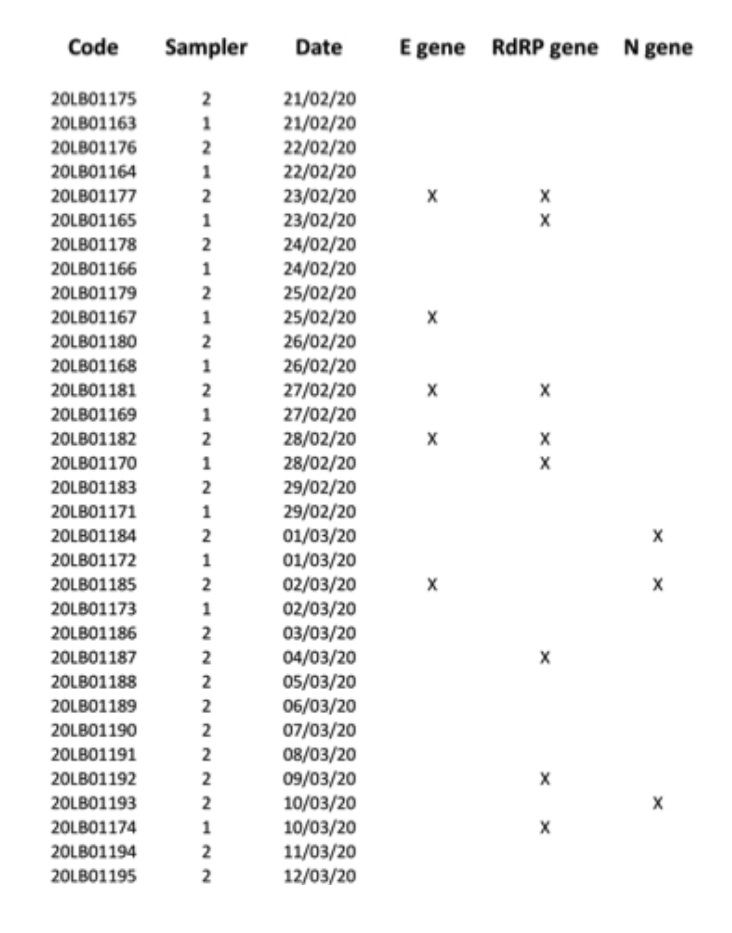
Aby uniknąć wyczerpania się dostępnego, ograniczonego materiału do pobierania próbek, pozostałe wyekstrahowane RNA dostarczono do lokalnego Szpitala Uniwersyteckiego (jednego z ośrodków klinicznych upoważnionych przez rząd włoski do przeprowadzania testów diagnostycznych SARS-CoV-2), aby wykonać drugi równoległy test ślepy. To drugie laboratorium kliniczne przetestowało 34 ekstrakcje RNA dla genów E, N i RdRP, zgłaszając 7 pozytywnych wyników dla co najmniej jednego z trzech genów markerowych, przy czym pozytywność potwierdzono oddzielnie dla wszystkich trzech markerów (ryc. 2). Ze względu na charakter próbki i biorąc pod uwagę, że pobieranie próbek nie zostało przeprowadzone w celach diagnostyki klinicznej, ale w celu przeprowadzenia testów zanieczyszczenia środowiska (biorąc również pod uwagę, że filtry były przechowywane przez co najmniej cztery tygodnie przed poddaniem ich analizom genetyki molekularnej, ponieważw wyniku włoskiego zamknięcia) możemy potwierdzić, że rozsądnie wykazaliśmy obecność wirusowego RNA SARS-CoV-2 poprzez wykrycie wysoce specyficznego „genu RtDR” na 8 filtrach. Jednak z powodu braku dodatkowych materiałów z filtrów nie byliśmy w stanie powtórzyć wystarczającej liczby testów, aby wykazać dodatni wynik dla wszystkich 3 markerów molekularnych jednocześnie.
To pierwszy wstępny dowód na to, że RNA SARS-CoV-2 może być obecne w cząstkach stałych na zewnątrz, co sugeruje, że w warunkach stabilności atmosferycznej i wysokich stężeń PM, SARS-CoV-2 może tworzyć skupiska z zewnętrznymi PM i – poprzez zmniejszenie ich współczynnika dyfuzji – zwiększać trwałość wirusa w atmosferze. Dalsze potwierdzenia tego wstępnegodowody są w toku i powinny obejmować ocenę w czasie rzeczywistym żywotności SARS-CoV-2, a także jego zjadliwości po zaadsorbowaniu na cząstkach stałych. Obecnie nie można poczynić żadnych założeń dotyczących korelacji między obecnością wirusa na PM a postępem epidemii COVID-19. Inne kwestie, które należy szczegółowo omówić, to średnie stężenia PM ostateczniewymagane do potencjalnego „efektu wzmocnienia” zakażenia (w przypadku potwierdzenia, że PM może działać jako „nośnik” dla jąder wirusowych kropelek) lub nawet teoretycznej możliwości uodpornienia w wyniku ekspozycji na minimalne dawki przy niższych progach PM.
Rys.1 Krzywe amplifikacji genów E (A) i RdRP (B): zielone linie oznaczają testowane filtry; linie krzyżoweprzedstawia ekstrakcje filtrów referencyjnych; czerwone linie przedstawiają amplifikację próbek dodatnich.
Rys. 2. Wyniki pozytywne (oznaczone X) dla genów E, N i RdRP uzyskane dla wszystkich 34 próbek PM10filtry testowane w drugiej analizie równoległej.
 Leonardo Setti1, Fabrizio Passarini2, Gianluigi De Gennaro3, Pierluigi Barbieri4, Maria Grazia Perrone5, Massimo Borelli6, Jolanda Palmisani3, Alessia Di Gilio3, Valentina Torboli6, Alberto Pallavicini6, Maurizio Ruscio7, Prisco Piscitelli8, Alessandro Miani8,9
Leonardo Setti1, Fabrizio Passarini2, Gianluigi De Gennaro3, Pierluigi Barbieri4, Maria Grazia Perrone5, Massimo Borelli6, Jolanda Palmisani3, Alessia Di Gilio3, Valentina Torboli6, Alberto Pallavicini6, Maurizio Ruscio7, Prisco Piscitelli8, Alessandro Miani8,9
1. Katedra Chemii Przemysłowej, Uniwersytet w Bolonii, Viale del Risorgimento – 4, I-40136, Bolonia, Włochy
e-mail: leonardo.setti@unibo.it
2. Międzywydziałowe Centrum Badań Przemysłowych „Źródła odnawialne, środowisko, wzrost niebieski, energia”,
University of Bologna, Rimini, Italy e-mail: fabrizio.passarini@unibo.it
3. Wydział Biologii, Uniwersytet „Aldo Moro” w Bari, Bari, Włochy
e-mail: gianluigi.degennaro@uniba.it; alessia.digilio@uniba.it; jolanda.palmisani@uniba.it
4. Wydział Nauk Chemicznych i Farmaceutycznych, Uniwersytet w Trieście, Triest, Włochy
e-mail: barbierp@units.it
5. Dział Badań Środowiskowych, TCR TECORA, Mediolan, Włochy
e-mail: mariagrazia.perrone@tcrtecora.com
6. Wydział Nauk o Życiu – Uniwersytet w Trieście, Triest, Włochy
e-mail: borelli@units.it; torboli@units.it; pallavic@units.it
7. Oddział Medycyny Laboratoryjnej, Szpital Uniwersytecki Giuliano Isontina (ASU GI), Triest, Włochy
email: maurizio.ruscio@asugi.sanita.fvg.it
8. Włoskie Towarzystwo Medycyny Środowiskowej (SIMA), Mediolan, Włochy
e-mail: priscofreedom@hotmail.com; alessandro.miani@unimi.it
9. Katedra Nauki o Środowisku i Polityki, Uniwersytet w Mediolanie, Mediolan, Włochy
e-mail: priscofreedom@hotmail.com; alessandro.miani@unimi.it
Autor korespondencyjny:
Leonardo Setti, Department of Industrial Chemistry, University of Bologna Viale del Risorgimento 4, 40136, Bologna, Italy; e-mail: leonardo.setti@unibo.it
Odniesienia
1. Światowa Organizacja Zdrowia, Sposoby transmisji wirusa powodującego COVID-19: implikacje dla zaleceń dotyczących środków ostrożności IPC, Omówienie naukowe; dostępne na stronie: https://www.who.int/newsroom/commentaries/detail/modes-of-transmission-of-virus-causing-covid-19-implications-for-ipcprecaution-recommendations (29 marca 2020 r.)
2. Włoskie Ministerstwo Zdrowia, codzienny biuletyn dotyczący epidemii Covid-19 we Włoszech, dostępny na stronie http://www.salute.gov.it/imgs/C_17_notizie_4451_0_file.pdf
3. EEA, Europejska Agencja Środowiska, Raport na temat jakości powietrza w Europie w 2019 r.; nr 10/2019; Europejska Agencja Środowiska: Kopenhaga, Dania, dostępny pod adresem: https://www.eea.europa.eu/publications/airquality-in-europe-2019
4. Xiao Wu, Rachel C. Nethery, M. Benjamin Sabath, Danielle Braun, Francesca Dominici, Narażenie na zanieczyszczenie powietrza i śmiertelność z powodu COVID-19 w Stanach Zjednoczonych, dostępne na stronie: https://projects.iq.harvard.edu/files/covid-pm/files/pm_and_covid_mortality.pdf
5. Włoskie Towarzystwo Medycyny Środowiskowej (SIMA), Dokument stanowiska Cząstki stałe i COVID-19,
dostępne na stronie: http://www.simaonlus.it/wpsima/wp-content/uploads/2020/03/COVID_19_positionpaper_ENG.pdf
6. Setti L., Passarini F., De Gennaro G., Barbieri P., Perrone MG, Piazzalunga A., Borelli M., Palmisani J., Di Gilio A, Piscitelli P, Miani A., Czy istnieje prawdopodobna rola cząstek stałych w rozprzestrzenianiu się COVID-19 w północnych Włoszech?, BMJ Rapid Responses, 8 kwietnia 2020 r., dostępne pod adresem: https://www.bmj.com/content/368/bmj.m1103/rapid-responses
7. Sedlmaier, N., Hoppenheidt, K., Krist, H., Lehmann, S., Lang, H., Buttner, M. Generowanie drobnych cząstek kałowych (PM2,5) zanieczyszczonych wirusem grypy ptaków (AIV): wykrywanie genomu i zakaźności oraz obliczanie imisji. Veterinary Microbiology. 139, 156-164 (2009)
8. Zhao, Y., Richardson, B., Takle, E., Chai, L., Schmitt, D., Win, H. Transmisja drogą powietrzną mogła odegrać rolę w rozprzestrzenianiu się ognisk wysoce zjadliwej grypy ptaków w 2015 r. w Stanach Zjednoczonych. Sci Rep. 9, 11755. https://doi.org/10.1038/s41598-019-47788-z (2019)
9. Ma, Y., Zhou, J., Yang, S., Zhao, Y., Zheng, X. Ocena wpływu zdarzeń pyłowych na występowanie odry w zachodnich Chinach. Atmospheric Environment. 157, 1-9 (2017)
10. Sorensen, JH, Mackay, DKJ, Jensen, C. Ø., Donaldson, AI Zintegrowany model przewidywania rozprzestrzeniania się wirusa pryszczycy w atmosferze Epidemiol. Infect., 124, 577–590 (2000)
11. Glostera, J., Alexandersen, S. Nowe kierunki: transmisja drogą powietrzną wirusa pryszczycy w środowisku atmosferycznym, 38 (3), 503-505 (2004)
12. Reche, I., D'Orta, G., Mladenov, N., Winget, DM, Suttle, CA Tempa osadzania się wirusów i bakterii powyżej warstwy granicznej atmosfery. Czasopismo ISME. 12, 1154-1162 (2018)
13. Qin, N., Liang, P., Wu, C., Wang, G., Xu, Q., Xiong, X., Wang, T., Zolfo, M., Segata, N., Qin, H., Knight, R., Gilbert, JA, Zhu, TF Longitudinalne badanie mikrobiomu związanego z cząstkami stałymi w megamieście. Genome Biology. 21, 55 (2020)
14. Zhao, Y., Richardson, B., Takle, E., Chai, L., Schmitt, D., Win, H. Transmisja drogą powietrzną może mieć
odegrał rolę w rozprzestrzenianiu się ognisk wysoce zjadliwej grypy ptaków w Stanach Zjednoczonych w 2015 r. Sci
Rep. 9, 11755. https://doi.org/10.1038/s41598-019-47788-z (2019)
15. Ma, Y., Zhou, J., Yang, S., Zhao, Y., Zheng, X. Ocena wpływu zdarzeń pyłowych na występowanie odry w zachodnich Chinach. Atmospheric Environment. 157, 1-9 (2017)
16. Jiang, W., Laing, P., Wang, B., Fang, J., Lang, J., Tian, G., Jiang, J., Zhu, TF Zoptymalizowana ekstrakcja DNA i sekwencjonowanie metagenomiczne społeczności mikroorganizmów unoszących się w powietrzu. Nat. Protoc. 10, 768-779 (2015)
17. Pan, M., Lednicky, JA, Wu, C.-Y., Gromadzenie, określanie wielkości cząstek i wykrywanie wirusów przenoszonych drogą powietrzną. Journal of Applied Microbiology, 127, 1596-1611 (2019)
18. Zymoresearch Ldt, opis produktu, dostępny na stronie: https://www.zymoresearch.com/products/quick-rnafecal-soil-microbe microprep-kit
19. Quantabio Ltd, opis produktu dostępny na stronie: https://www.quantabio.com/qscript-xlt-1-steprt-qpcr-toughmix
20. Corman, VM, Landt, O., Kaiser, M., Molenkamp, R., Meijer, A., Chu, DK i Mulders, DG (2020).
Wykrywanie nowego koronawirusa 2019 (2019-nCoV) metodą RT-PCR w czasie rzeczywistym. Eurosurveillance, 25(3), dostępne na stronie: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6988269/
Oryginał: https://doi.org/10.1101/2020.04.15.20065995
Czas publikacji: 18-kwi-2020








