La sindrome respiratoria acuta grave nota come malattia COVID-19 – dovuta al virus SARS-CoV-2 – è nota per diffondersi attraverso le goccioline respiratorie e i contatti stretti.[1] Il peso del COVID-19 è stato estremamente grave in Lombardia e Pianura Padana (Italia settentrionale),[2] un'area caratterizzata da elevate concentrazioni di particolato, già noto per produrre effetti negativi sulla salute umana.[3] I dati regionali disponibili per l'Italia alla data del 12 aprile mostrano che circa il 30% delle persone attualmente positive vive ancora in Lombardia (circa il 40% se si considerano i casi totali confermati dall'inizio dell'epidemia), seguita da Emilia Romagna (13,5%), Piemonte (10,5%) e Veneto (10%).[2] Queste quattro regioni della Pianura Padana rappresentano l'80% del totale dei decessi registrati in Italia e il 65% dei ricoveri in terapia intensiva.[2]
Una ricerca condotta dalla Harvard School of Public Health sembra confermare un’associazione tra aumenti delle concentrazioni di PM e tassi di mortalità dovuti al COVID-19 negli USA[4] In precedenti comunicazioni, abbiamo ipotizzato la possibilità che il virus SARS-CoV-2 potesse essere presente sul particolato (PM) durante la diffusione dell’infezione,[5,6] in linea con le evidenze già
disponibile per altri virus.[7-15] Tuttavia, la questione del microbioma associato al PM presente nell’aria, soprattutto negli ambienti urbani, rimane in gran parte poco studiata,[16] e – al momento – nessuno ha ancora condotto studi sperimentali specificamente volti a confermare o escludere la presenza del SARS-CoV-2 sul PM.
Presentiamo qui i primi risultati delle analisi che abbiamo eseguito su 34 campioni di PM10 atmosferico/esterno provenienti da un sito industriale della provincia di Bergamo, raccolti con due diversi campionatori d'aria nell'arco di un periodo continuativo di 3 settimane, dal 21 febbraio al 13 marzo.
Seguendo la metodologia descritta da Pan et al. nel 2019 (per la raccolta, la granulometria e il rilevamento di virus aerodispersi),[17] i campioni di PM sono stati raccolti su filtri in fibra di quarzo utilizzando un campionatore d'aria gravimetrico a basso volume (38,3 l/min per 23 h), conforme al metodo di riferimento EN12341:2014 per il monitoraggio del PM10. Il particolato è stato intrappolato sui filtri con una concentrazione tipica del 99,9%.ritenzione di aerosol, opportunamente conservato e consegnato al laboratorio di Genomica Applicata e Comparata dell'Università di Trieste. Data la natura "ambientale" del campione, presumibilmente ricco di inibitori delle DNA polimerasi, si è proceduto all'estrazione dell'RNA utilizzando il kit Quick RNA fecal soil microbie adattato alla tipologia dei filtri.[18] Il filtro è stato arrotolato per metà, con il lato superiore rivolto verso l'interno,in una provetta in polipropilene da 5 ml, insieme alle biglie fornite nel kit. Dal ml iniziale di tampone di lisi, siamo stati in grado di ottenere circa 400 ul di soluzione, che è stata poi processata come definito dai protocolli standard, ottenendo un eluato finale di 15 ul. Successivamente, 5 ul sono stati utilizzati per il test SARS-CoV-2. Data la particolare origine del campione, è stato utilizzato il qScript XLT 1-Step RT-qPCR ToughMix.[19] I sistemi di amplificazione erano quelli del protocollo sviluppato da Corman et al, pubblicato sul sito web dell'OMS [20].
Il test era esplicitamente mirato a confermare o escludere la presenza dell'RNA del SARS-CoV-2 sul particolato. La prima analisi ha utilizzato il "gene E" come marcatore molecolare e ha prodotto un risultato positivo impressionante su 15 filtri su 16, anche se, come prevedibile, il Ct era compreso tra 36 e 38 cicli.
Successivamente, abbiamo replicato l’analisi su 6 dei filtri positivi (già positivi al “gene E”) utilizzando come marcatore molecolare il “gene RtDR” – altamente specifico per SARS-CoV-2 – raggiungendo 5 risultati significativi di positività; sono stati eseguiti con successo anche test di controllo per escludere false positività (Fig. 1).
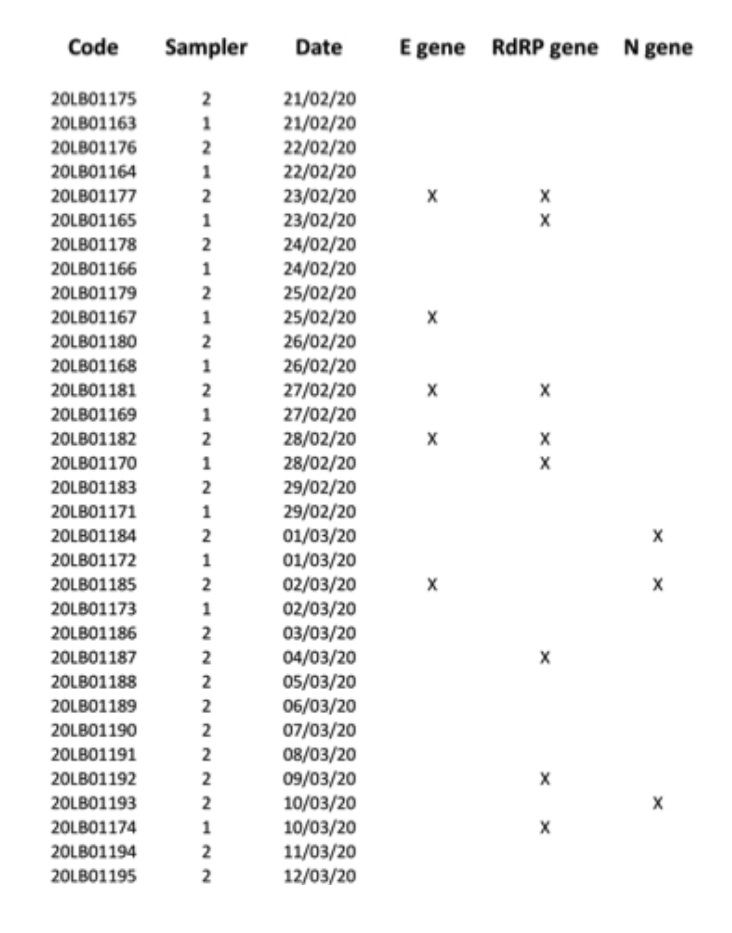
Per evitare l'esaurimento del materiale di campionamento disponibile, i restanti RNA estratti sono stati consegnati all'Azienda Ospedaliero-Universitaria locale (uno dei centri clinici autorizzati dal Governo italiano per i test diagnostici per SARS-CoV-2), al fine di eseguire un secondo test cieco parallelo. Questo secondo laboratorio clinico ha testato 34 estrazioni di RNA per i geni E, N e RdRP, riportando 7 risultati positivi per almeno uno dei tre geni marcatori, con positività confermata separatamente per tutti e tre i marcatori (Fig. 2). Data la natura del campione, e considerando che il campionamento non è stato effettuato per scopi di diagnostica clinica ma per test di inquinamento ambientale (tenendo anche conto che i filtri sono stati conservati per almeno quattro settimane prima di essere sottoposti ad analisi di genetica molecolare, comeA seguito del lockdown italiano, possiamo confermare di aver ragionevolmente dimostrato la presenza di RNA virale SARS-CoV-2 rilevando il gene RtDR altamente specifico su 8 filtri. Tuttavia, a causa della mancanza di materiale aggiuntivo dai filtri, non siamo stati in grado di ripetere un numero sufficiente di test per dimostrare la positività per tutti e 3 i marcatori molecolari contemporaneamente.
Questa è la prima evidenza preliminare che l'RNA di SARS-CoV-2 può essere presente sul particolato atmosferico esterno, suggerendo che, in condizioni di stabilità atmosferica e di elevate concentrazioni di PM, SARS-CoV-2 potrebbe creare cluster con il PM esterno e, riducendone il coefficiente di diffusione, aumentare la persistenza del virus in atmosfera. Ulteriori conferme di questa evidenza preliminareLe prove sono in corso e dovrebbero includere una valutazione in tempo reale della vitalità del SARS-CoV-2 e della sua virulenza quando adsorbito sul particolato. Al momento, non è possibile formulare ipotesi sulla correlazione tra la presenza del virus sul PM e la progressione dell'epidemia di COVID-19. Altre questioni da affrontare specificamente sono le concentrazioni medie di PM alla finenecessaria per un potenziale “effetto boost” del contagio (nel caso in cui venga confermato che il PM possa agire da “vettore” per i nuclei virali delle goccioline), o addirittura la possibilità teorica di immunizzazione conseguente a esposizioni a dosi minime a soglie inferiori di PM.
Fig.1 Curve di amplificazione dei geni E (A) e RdRP (B): le linee verdi rappresentano i filtri testati; le linee incrociaterappresenta le estrazioni del filtro di riferimento; le linee rosse rappresentano l'amplificazione dei campioni positivi.
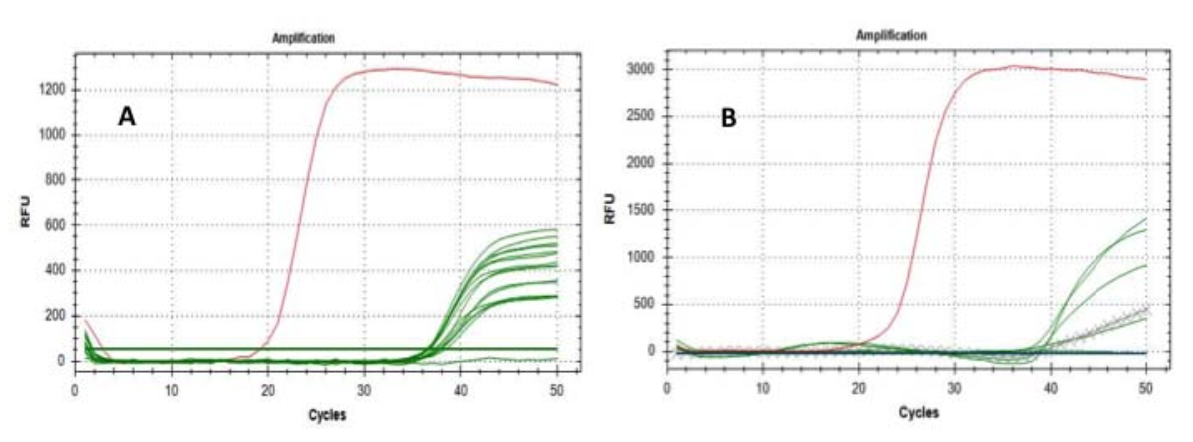
Fig.2. Risultati positivi (contrassegnati con X) per i geni E, N e RdRP ottenuti per tutti i 34 campioni di PM10filtri testati nelle seconde analisi parallele.
 Leonardo Setti1, Fabrizio Passarini2, Gianluigi De Gennaro3, Pierluigi Barbieri4, Maria Grazia Perrone5, Massimo Borelli6, Jolanda Palmisani3, Alessia Di Gilio3, Valentina Torboli6, Alberto Pallavicini6, Maurizio Ruscio7, Prisco Piscitelli8, Alessandro Miani8,9
Leonardo Setti1, Fabrizio Passarini2, Gianluigi De Gennaro3, Pierluigi Barbieri4, Maria Grazia Perrone5, Massimo Borelli6, Jolanda Palmisani3, Alessia Di Gilio3, Valentina Torboli6, Alberto Pallavicini6, Maurizio Ruscio7, Prisco Piscitelli8, Alessandro Miani8,9
1. Dip. Chimica Industriale, Università di Bologna, Viale del Risorgimento – 4, I-40136, Bologna, Italia
e-mail: leonardo.setti@unibo.it
2. Centro Interdipartimentale di Ricerca Industriale “Fonti Rinnovabili, Ambiente, Crescita Blu, Energia”,
University of Bologna, Rimini, Italy e-mail: fabrizio.passarini@unibo.it
3. Dipartimento di Biologia, Università “Aldo Moro” di Bari, Bari, Italia
e-mail: gianluigi.degennaro@uniba.it; alessia.digilio@uniba.it; jolanda.palmisani@uniba.it
4. Dipartimento di Scienze Chimiche e Farmaceutiche, Università di Trieste, Trieste, Italia
e-mail: barbierp@units.it
5. Divisione di Ricerca Ambientale, TCR TECORA, Milano, Italia
e-mail: mariagrazia.perrone@tcrtecora.com
6. Dipartimento di Scienze della Vita – Università di Trieste, Trieste, Italia
e-mail: borelli@units.it; torboli@units.it; pallavic@units.it
7. Divisione di Medicina di Laboratorio, Azienda Ospedaliera Universitaria Giuliano Isontina (ASU GI), Trieste, Italia
email: maurizio.ruscio@asugi.sanita.fvg.it
8. Società Italiana di Medicina Ambientale (SIMA), Milano, Italia
e-mail: priscofreedom@hotmail.com; alessandro.miani@unimi.it
9. Dipartimento di Scienze Ambientali e Politica, Università degli Studi di Milano, Milano, Italia
e-mail: priscofreedom@hotmail.com; alessandro.miani@unimi.it
Autore corrispondente:
Leonardo Setti, Department of Industrial Chemistry, University of Bologna Viale del Risorgimento 4, 40136, Bologna, Italy; e-mail: leonardo.setti@unibo.it
Riferimenti
1. Organizzazione Mondiale della Sanità, Modalità di trasmissione del virus che causa COVID-19: implicazioni per le raccomandazioni precauzionali dell'IPC, Nota scientifica; disponibile all'indirizzo: https://www.who.int/newsroom/commentaries/detail/modes-of-transmission-of-virus-causing-covid-19-implications-for-ipcprecaution-recommendations (29 marzo 2020)
2. Ministero della Salute italiano, bollettino quotidiano sull'epidemia di Covid-19 in Italia, disponibile all'indirizzo http://www.salute.gov.it/imgs/C_17_notizie_4451_0_file.pdf
3. AEA, Agenzia europea dell'ambiente, Rapporto sulla qualità dell'aria in Europa 2019; n. 10/2019; Agenzia europea dell'ambiente: Copenaghen, Danimarca, disponibile all'indirizzo: https://www.eea.europa.eu/publications/airquality-in-europe-2019
4. Xiao Wu, Rachel C. Nethery, M. Benjamin Sabath, Danielle Braun, Francesca Dominici, Esposizione all'inquinamento atmosferico e mortalità da COVID-19 negli Stati Uniti, disponibile all'indirizzo: https://projects.iq.harvard.edu/files/covid-pm/files/pm_and_covid_mortality.pdf
5. Società Italiana di Medicina Ambientale (SIMA), Position Paper Particulate Matter e COVID-19,
disponibile all'indirizzo: http://www.simaonlus.it/wpsima/wp-content/uploads/2020/03/COVID_19_positionpaper_ITA.pdf
6. Setti L., Passarini F., De Gennaro G., Barbieri P., Perrone MG, Piazzalunga A., Borelli M., Palmisani J., Di Gilio A, Piscitelli P, Miani A., Is there a Plausible Role for Particulate Matter in the spreading of COVID-19 in Northern Italy?, BMJ Rapid Responses, 8 aprile 2020, disponibile all'indirizzo: https://www.bmj.com/content/368/bmj.m1103/rapid-responses
7. Sedlmaier, N., Hoppenheidt, K., Krist, H., Lehmann, S., Lang, H., Buttner, M. Generazione di particolato fine fecale (PM2.5) contaminato dal virus dell'influenza aviaria (AIV): genoma, rilevamento dell'infettività e calcolo dell'immissione. Microbiologia Veterinaria. 139, 156-164 (2009)
8. Zhao, Y., Richardson, B., Takle, E., Chai, L., Schmitt, D., Win, H. La trasmissione aerea potrebbe aver giocato un ruolo nella diffusione delle epidemie di influenza aviaria ad alta patogenicità del 2015 negli Stati Uniti. Sci Rep. 9, 11755. https://doi.org/10.1038/s41598-019-47788-z (2019)
9. Ma, Y., Zhou, J., Yang, S., Zhao, Y., Zheng, X. Valutazione dell'impatto degli eventi di polvere sull'incidenza del morbillo nella Cina occidentale. Atmospheric Environment. 157, 1-9 (2017)
10. Sorensen, JH, Mackay, DKJ, Jensen, C. Ø., Donaldson, AI Un modello integrato per prevedere la diffusione atmosferica del virus dell'afta epizootica Epidemiol. Infect., 124, 577–590 (2000)
11. Glostera, J., Alexandersen, S. Nuove direzioni: trasmissione aerea del virus della febbre aftosa nell'ambiente atmosferico, 38 (3), 503-505 (2004)
12. Reche, I., D'Orta, G., Mladenov, N., Winget, DM, Suttle, CA Tassi di deposizione di virus e batteri al di sopra dello strato limite atmosferico. The ISME Journal. 12, 1154-1162 (2018)
13. Qin, N., Liang, P., Wu, C., Wang, G., Xu, Q., Xiong, X., Wang, T., Zolfo, M., Segata, N., Qin, H., Knight, R., Gilbert, JA, Zhu, TF Indagine longitudinale del microbioma associato al particolato in una megalopoli. Genome Biology. 21, 55 (2020)
14. Zhao, Y., Richardson, B., Takle, E., Chai, L., Schmitt, D., Win, H. La trasmissione aerea può avere
ha avuto un ruolo nella diffusione delle epidemie di influenza aviaria altamente patogena del 2015 negli Stati Uniti. Sci
Rappresentante 9, 11755. https://doi.org/10.1038/s41598-019-47788-z (2019)
15. Ma, Y., Zhou, J., Yang, S., Zhao, Y., Zheng, X. Valutazione dell'impatto degli eventi di polvere sull'incidenza del morbillo nella Cina occidentale. Atmospheric Environment. 157, 1-9 (2017)
16. Jiang, W., Laing, P., Wang, B., Fang, J., Lang, J., Tian, G., Jiang, J., Zhu, TF Estrazione ottimizzata del DNA e sequenziamento metagenomico di comunità microbiche aerodisperse. Nat. Protoc. 10, 768-779 (2015)
17. Pan, M., Lednicky, JA, Wu, C.-Y., Raccolta, dimensionamento delle particelle e rilevamento di virus aerodispersi. Journal of Applied Microbiology, 127, 1596-1611 (2019)
18. Zymoresearch Ldt, descrizione del prodotto, disponibile su: https://www.zymoresearch.com/products/quick-rnafecal-soil-microbe microprep-kit
19. Quantabio Ltd, descrizione del prodotto, disponibile su: https://www.quantabio.com/qscript-xlt-1-steprt-qpcr-toughmix
20. Corman, VM, Landt, O., Kaiser, M., Molenkamp, R., Meijer, A., Chu, DK, & Mulders, DG (2020).
Rilevamento del nuovo coronavirus 2019 (2019-nCoV) mediante RT-PCR in tempo reale. Eurosurveillance, 25(3), disponibile all'indirizzo: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6988269/
Originale: https://doi.org/10.1101/2020.04.15.20065995
Data di pubblicazione: 18 aprile 2020








