Ir atzīts, ka SARS-CoV-2 vīrusa izraisīts smags akūts respiratorais sindroms, kas pazīstams kā COVID-19 slimība, izplatās elpceļu pilienu un cieša kontakta ceļā.[1]Covid-19 izraisītais slogs bija ārkārtīgi smags Lombardijā un Po ielejā (Ziemeļitālijā)[2], kur ir liela daļiņu koncentrācija, kas jau ir zināma kā negatīva ietekme uz cilvēku veselību.[3]Reģionālie dati, kas pieejami par Itāliju 12. aprīļa datumā, liecina, ka aptuveni 30% no pašlaik pozitīviem cilvēkiem joprojām dzīvo Lombardijā (apmēram 40%, ja ņem vērā kopējos gadījumus, kas apstiprināti kopš epidēmijas sākuma), kam seko Emīlija-Romanja (13,5%). , Pjemonta (10,5 %) un Veneto (10 %).[2]Šie četri Po ielejas reģioni veido 80% no visiem Itālijā reģistrētajiem nāves gadījumiem un 65% no uzņemtajiem intensīvās terapijas nodaļās.[2]
Šķiet, ka Hārvardas Sabiedrības veselības skolas veiktais pētījums apstiprina saistību starp PM koncentrācijas palielināšanos un mirstības līmeni Covid-19 dēļ ASV[4] Iepriekšējos paziņojumos esam izvirzījuši hipotēzi, ka SARS-CoV-2. vīruss var atrasties uz daļiņām (PM) infekcijas izplatīšanās laikā,[5,6] atbilstoši pierādījumiem
ir pieejams citiem vīrusiem.[7-15] Tomēr jautājums par PM saistīto mikrobiomu gaisā, jo īpaši pilsētvidē, joprojām ir pārāk maz pētīts[16], un pašlaik neviens vēl nav veicis eksperimentālus pētījumus, kas būtu īpaši vērsti uz to. apstiprinot vai izslēdzot SARS-CoV-2 klātbūtni PM.
Šeit mēs piedāvājam pirmos analīžu rezultātus, ko esam veikuši 34 PM10 PM10 paraugiem no āra/gaisa PM10 no rūpnieciskās vietas Bergamo provincē, kas savākti ar diviem dažādiem gaisa paraugu ņēmējiem nepārtrauktā 3 nedēļu periodā no 21. februāra līdz martam. 13.
Pēc Pan et al. aprakstītās metodoloģijas.2019. gadā (gaisa vīrusu savākšanai, daļiņu izmēra noteikšanai un noteikšanai)[17] PM paraugi tika savākti uz kvarca šķiedras filtriem, izmantojot maza tilpuma gravimetrisko gaisa paraugu ņemšanas ierīci (38,3 l/min 23 stundas), kas atbilst standartmetodi EN12341. :2014 PM10 monitoringam.Cietās daļiņas tika notvertas uz filtriem ar 99,9% tipiskuaerosola aizture, pareizi uzglabāta un nogādāta Triestes universitātes Lietišķās un salīdzinošās genomikas laboratorijā.Ņemot vērā parauga “vides” raksturu, kas, iespējams, ir bagāts ar DNS polimerāzes inhibitoriem, mēs turpinājām RNS ekstrakciju, izmantojot Quick RNA fekālo augsnes mikrobu komplektu, kas pielāgots filtru veidam.[18]Pusfiltrs tika sarullēts ar augšējo pusi uz iekšu,5 ml polipropilēna mēģenē kopā ar komplektā iekļautajām lodītēm.No sākotnējā 1 ml lizabufera mēs varējām iegūt aptuveni 400 ul šķīduma, kas pēc tam tika apstrādāts, kā noteikts standarta protokolos, kā rezultātā gala eluāts bija 15 ul.Pēc tam SARS-CoV-2 testēšanai tika izmantoti 5 ul.Ņemot vērā parauga īpašo izcelsmi, tika izmantots qScript XLT 1-Step RT-qPCR ToughMix.[19]Pastiprināšanas sistēmas bija protokolā, ko izstrādāja Corman et al, kas publicēts PVO tīmekļa vietnē [20].
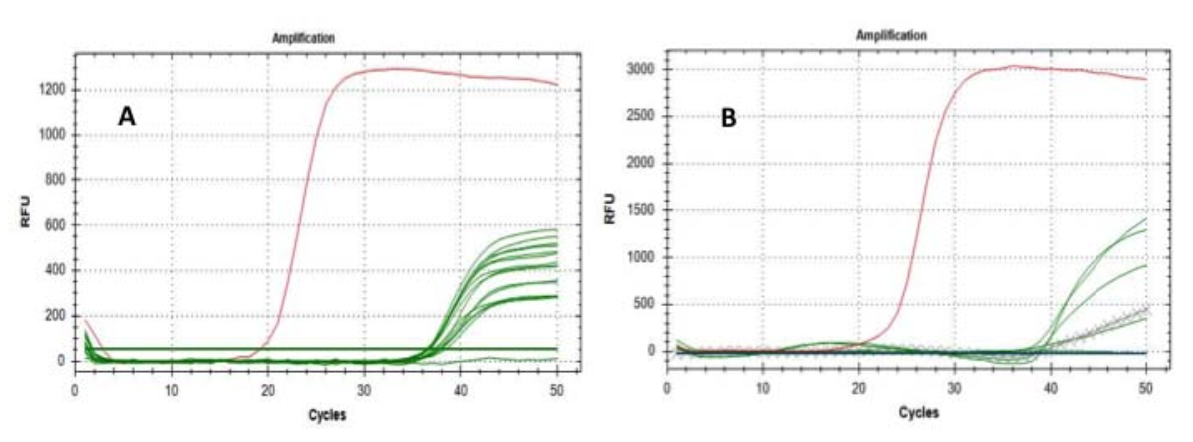
Testa mērķis bija skaidri apstiprināt vai izslēgt SARS-CoV-2 RNS klātbūtni uz daļiņām.Pirmajā analīzē tika izmantots “E gēns” kā molekulārais marķieris un tika iegūts iespaidīgs pozitīvs rezultāts 15 no 16 filtriem, pat ja, kā mēs varētu gaidīt, Ct bija no 36 līdz 38 cikliem.
Pēc tam mēs esam atkārtojuši analīzi uz 6 pozitīvajiem filtriem (jau pozitīvi uz "E gēnu"), izmantojot "RtDR gēnu" kā molekulāro marķieri, kas ir ļoti specifisks SARS-CoV-2, sasniedzot 5 nozīmīgus rezultātus. pozitivitāte;veiksmīgi tika veikti arī kontroles testi, lai izslēgtu viltus pozitivitāti (1. att.).
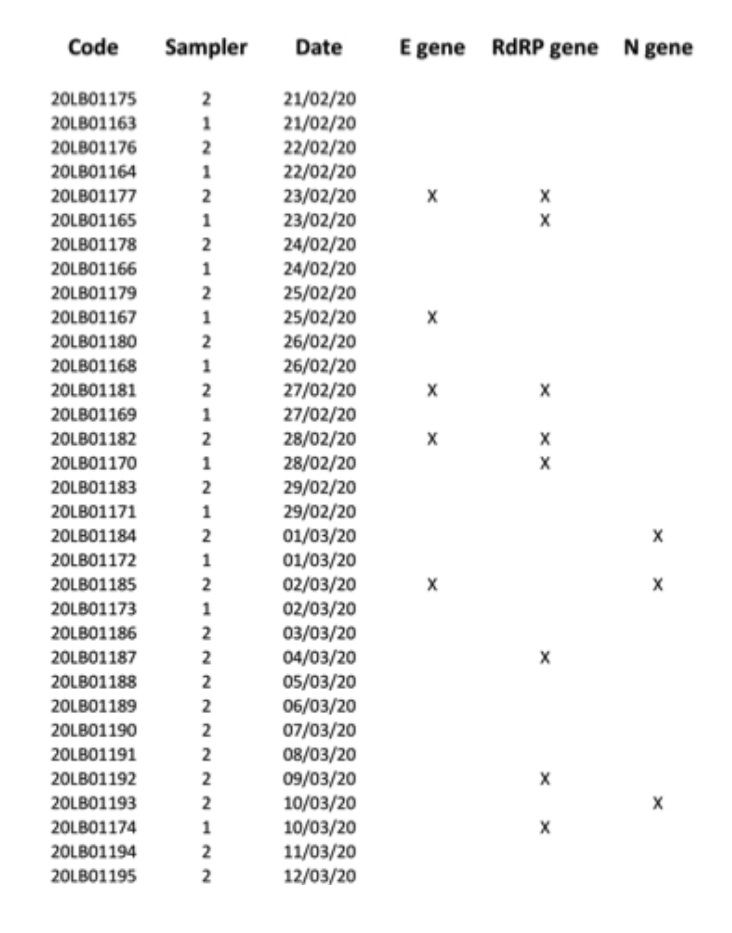
Lai izvairītos no ierobežotā pieejamā paraugu ņemšanas materiāla izbeigšanās, atlikušās ekstrahētās RNS tika nogādātas vietējā universitātes slimnīcā (vienā no klīniskajiem centriem, ko Itālijas valdība pilnvarojusi SARS-CoV-2 diagnostikas testiem), lai veiktu otru. paralēlais akls tests.Šī otrā klīniskā laboratorija pārbaudīja 34 RNS ekstrakcijas E, N un RdRP gēniem, ziņojot par 7 pozitīviem rezultātiem vismaz vienam no trim marķieru gēniem, un pozitivitāte tika atsevišķi apstiprināta visiem trim marķieriem (2. attēls).Ņemot vērā parauga raksturu un ņemot vērā to, ka paraugu ņemšana nav veikta klīniskās diagnostikas nolūkos, bet gan vides piesārņojuma pārbaudēm (ņemot vērā arī to, ka filtri tika uzglabāti vismaz četras nedēļas pirms molekulāro ģenētisko analīžu veikšanas, kā arīItālijas slēgšanas sekas), mēs varam apstiprināt, ka esam pamatoti pierādījuši SARS-CoV-2 vīrusa RNS klātbūtni, atklājot ļoti specifisku “RtDR gēnu” 8 filtros.Tomēr, tā kā no filtriem trūka papildu materiālu, mēs nevarējām atkārtot pietiekami daudz testu, lai vienlaikus parādītu pozitivitāti visiem 3 molekulārajiem marķieriem.
Šis ir pirmais sākotnējais pierādījums tam, ka SARS-CoV-2 RNS var atrasties uz āra daļiņām, tādējādi liekot domāt, ka atmosfēras stabilitātes apstākļos un augstās PM koncentrācijās SARS-CoV-2 varētu radīt kopas ar āra PM un – samazinot to difūzijas koeficientu – uzlabo vīrusa noturību atmosfērā.Papildu apstiprinājumi šim iepriekšējamPierādījumi turpinās, un tajos jāietver reāllaika novērtējums par SARS-CoV-2 vitalitāti, kā arī tā virulenci, kad tā adsorbēta uz daļiņām.Pašlaik nevar izdarīt nekādus pieņēmumus par korelāciju starp vīrusa klātbūtni PM un Covid-19 uzliesmojuma progresēšanu.Citi jautājumi, kas īpaši jārisina, ir PM vidējā koncentrācijanepieciešama infekcijas potenciālajam “pastiprināšanas efektam” (ja tiek apstiprināts, ka PM varētu darboties kā vīrusa pilienu kodolu “nesējs”) vai pat teorētiskai imunizācijas iespējai, kas izriet no minimālas devas iedarbības pie zemākiem PM sliekšņiem. .
1. att. E (A) un RdRP gēnu (B) amplifikācijas līknes: zaļās līnijas attēlo pārbaudītos filtrus;krustojuma līnijasapzīmē atsauces filtru ekstrakcijas;sarkanās līnijas apzīmē pozitīvo paraugu pastiprināšanos.
2. att.Pozitīvi rezultāti (apzīmēti ar X) E, N un RdRP gēniem, kas iegūti visiem 34 PM10 paraugiemotrajā paralēlajā analīzē pārbaudītajiem filtriem.
 Leonardo Setti1, Fabricio Pasarīni2, Džanluidži De Gennaro3, Pjerluidži Barbjēri4, Marija Gracija Perrone5, Masimo Borelli6, Jolanda Palmisani3, Alesija Di Žilio3, Valentīna Torboli6, Alberto Palavičīni6, Mauricio Ruscio7, Prisko A,9 Piscitelli8,8
Leonardo Setti1, Fabricio Pasarīni2, Džanluidži De Gennaro3, Pjerluidži Barbjēri4, Marija Gracija Perrone5, Masimo Borelli6, Jolanda Palmisani3, Alesija Di Žilio3, Valentīna Torboli6, Alberto Palavičīni6, Mauricio Ruscio7, Prisko A,9 Piscitelli8,8
1. Boloņas Universitātes Rūpnieciskās ķīmijas nodaļa, Viale del Risorgimento – 4, I-40136, Boloņa, Itālija
e-mail: leonardo.setti@unibo.it
2. Starpresoru rūpniecisko pētījumu centrs “Atjaunojamie avoti, vide, zilā izaugsme, enerģētika”,
University of Bologna, Rimini, Italy e-mail: fabrizio.passarini@unibo.it
3. Bioloģijas katedra, Bari universitāte “Aldo Moro”, Bari, Itālija
e-mail: gianluigi.degennaro@uniba.it; alessia.digilio@uniba.it; jolanda.palmisani@uniba.it
4. Ķīmijas un farmācijas zinātņu departaments, Triestes Universitāte, Trieste, Itālija
e-mail: barbierp@units.it
5. Vides pētījumu nodaļa, TCR TECORA, Milāna, Itālija
e-mail: mariagrazia.perrone@tcrtecora.com
6. Dzīvības zinātņu nodaļa – Triestes Universitāte, Trieste, Itālija
e-mail: borelli@units.it; torboli@units.it; pallavic@units.it
7. Laboratoriskās medicīnas nodaļa, Giuliano Isontina universitātes slimnīca (ASU GI), Trieste, Itālija
email: maurizio.ruscio@asugi.sanita.fvg.it
8. Itālijas Vides medicīnas biedrība (SIMA), Milāna, Itālija
e-mail: priscofreedom@hotmail.com; alessandro.miani@unimi.it
9. Milānas Universitātes Vides zinātnes un Poicy nodaļa, Milāna, Itālija
e-mail: priscofreedom@hotmail.com; alessandro.miani@unimi.it
Atbilstošais autors:
Leonardo Setti, Department of Industrial Chemistry, University of Bologna Viale del Risorgimento 4, 40136, Bologna, Italy; e-mail: leonardo.setti@unibo.it
Atsauces
1. Pasaules Veselības organizācija, Covid-19 izraisoša vīrusa pārnešanas veidi: ietekme uz IPC piesardzības ieteikumiem, Zinātniskā informācija;pieejams: https://www.who.int/newsroom/commentaries/detail/modes-of-transmission-of-virus-causing-covid-19-implications-for-ipcprecaution-recommendations (2020. gada 29. martā)
2. Itālijas Veselības ministrijas ikdienas biļetens par Covid-19 uzliesmojumu Itālijā, pieejams vietnē http://www.salute.gov.it/imgs/C_17_notizie_4451_0_file.pdf
3. EVA, Eiropas Vides aģentūra, Gaisa kvalitāte Eiropā 2019. gada ziņojums;Nr. 10/2019;Eiropas Vides aģentūra: Kopenhāgena, Dānija, pieejama: https://www.eea.europa.eu/publications/airquality-in-europe-2019
4. Xiao Wu, Rachel C. Nethery, M. Benjamin Sabath, Danielle Braun, Francesca Dominici, Pakļaušana gaisa piesārņojumam un mirstība no COVID-19 Amerikas Savienotajās Valstīs, pieejama vietnē https://projects.iq.harvard.edu/ files/covid-pm/files/pm_and_covid_mortality.pdf
5. Itālijas Vides medicīnas biedrība (SIMA), Position Paper Particulate Matter un Covid-19,
pieejams: http://www.simaonlus.it/wpsima/wp-content/uploads/2020/03/COVID_19_positionpaper_ENG.pdf
6. Setti L., Passarīni F., De Gennaro G., Barbieri P., Perrone MG, Piazzalunga A., Borelli M., Palmisani J., Di Gilio A, Piscitelli P, Miani A., Vai pastāv ticama loma par cietajām vielām COVID-19 izplatībā Ziemeļitālijā?, BMJ Rapid Responses, 2020. gada 8. aprīlis, pieejams vietnē: https://www.bmj.com/content/368/bmj.m1103/rapid-responses
7. Sedlmaier, N., Hoppenheidt, K., Krist, H., Lehmann, S., Lang, H., Buttner, M. Generation of avian influenza virus (AIV) contaminated fecal fine particulate material (PM2.5): genoma un infekciozitātes noteikšana un immisijas aprēķināšana.Veterinārā mikrobioloģija.139, 156-164 (2009)
8. Zhao, Y., Richardson, B., Takle, E., Chai, L., Schmitt, D., Win, H. Airborne transmisija varētu būt bijusi loma 2015. gada augsti patogēnas putnu gripas uzliesmojumu izplatībā. Savienotās Valstis.Sci Rep. 9, 11755. https://doi.org/10.1038/s41598-019-47788-z (2019)
9. Ma, Y., Zhou, J., Yang, S., Zhao, Y., Zheng, X. Novērtējums putekļu notikumu ietekmei uz masalu incidenci Ķīnas rietumos.Atmosfēras vide.157, 1-9 (2017)
10. Sorensen, JH, Mackay, DKJ, Jensen, C. Ø., Donaldson, AI Integrēts modelis, lai prognozētu mutes un nagu sērgas vīrusa Epidemiol izplatību atmosfērā.Infect., 124, 577–590 (2000)
11. Glostera, J., Alexandersen, S. New Directions: Airborne Transmission of Mutes un nagu slimību vīrusa atmosfēras vide, 38 (3), 503-505 (2004)
12. Reche, I., D'Orta, G., Mladenov, N., Winget, DM, Suttle, CA Vīrusu un baktēriju nogulsnēšanās ātrums virs atmosfēras robežslāņa.ISME žurnāls.12, 1154-1162 (2018)
13. Cjin, N., Liang, P., Wu, C., Wang, G., Xu, Q., Xiong, X., Wang, T., Zolfo, M., Segata, N., Qin, H. ., Knight, R., Gilbert, JA, Zhu, TF Garengriezuma mikrobioma apsekojums, kas saistīts ar cietajām daļiņām megapilsētā.Genoma bioloģija.21, 55 (2020)
14. Zhao, Y., Richardson, B., Takle, E., Chai, L., Schmitt, D., Win, H. Airborne pārraidei var būt
bija nozīme 2015. gada augsti patogēnās putnu gripas uzliesmojumu izplatībā Amerikas Savienotajās Valstīs.Sci
Rep. 9, 11755. https://doi.org/10.1038/s41598-019-47788-z (2019)
15. Ma, Y., Zhou, J., Yang, S., Zhao, Y., Zheng, X. Putekļu notikumu ietekmes uz masalu sastopamību Ķīnas rietumos.Atmosfēras vide.157, 1-9 (2017)
16. Jiang, W., Laing, P., Wang, B., Fang, J., Lang, J., Tian, G., Jiang, J., Zhu, TF Optimizēta DNS ekstrakcija un metagenomiskā sekvencēšana gaisā esošu mikrobu kopienu .Nat.Protok.10, 768-779 (2015)
17. Pan, M., Lednicky, JA, Wu, C.-Y., Savākšana, daļiņu lielums un noteikšana gaisā.Journal of Applied Microbiology, 127, 1596-1611 (2019)
18. Zymoresearch Ldt, produkta apraksts, pieejams: https://www.zymoresearch.com/products/quick-rnafecal-soil-microbe microprep-kit
19. Quantabio Ltd, produkta apraksts, pieejams: https://www.quantabio.com/qscript-xlt-1-steprt-qpcr-toughmix
20. Corman, VM, Landt, O., Kaiser, M., Molenkamp, R., Meijer, A., Chu, DK un Mulders, DG (2020).
2019. gada jaunā koronavīrusa (2019-nCoV) noteikšana ar reāllaika RT-PCR.Eurosurveillance, 25(3), pieejams: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6988269/
Oriģināls: https://doi.org/10.1101/2020.04.15.20065995
Publicēšanas laiks: 18.04.2020
