U sindromu respiratoriu agutu severu cunnisciutu cum'è malatia COVID-19 - per via di u virus SARS-CoV-2 - hè ricunnisciutu per sparghje per via di gocce respiratorii è cuntatti stretti.[1]U pesu di COVID-19 era estremamente severu in Lombardia è in u Po Valley (Italia di u Nordu),[2] una zona carattarizata da alta concentrazione di particulate, chì sò digià cunnisciute per pruduce effetti negativi nantu à a salute umana.[3]I figuri righjunali dispunibuli per l'Italia à a data di u 12 d'aprile mostranu chì circa 30% di e persone attualmente pusitive vivenu sempre in Lombardia (circa 40% se cunziddi i casi generali cunfirmati da u principiu di l'epidemie), seguitu da Emilia Romagna (13,5%). , Piemonte (10,5%), è Venetu (10%).[2]Queste quattru regioni di u Po Valley cuntanu l'80% di i morti totali registrati in Italia è u 65% di l'ingressu à l'Unità di Cura Intensiva.[2]
Una ricerca realizata da a Scola di Salute Pública di Harvard pare cunfirmà una associazione trà l'aumentu di a concentrazione di PM è i tassi di mortalità per via di COVID-19 in i Stati Uniti [4] In cumunicazioni precedenti, avemu ipotizatu a pussibilità chì SARS-CoV-2. U virus puderia esse presente nantu à a materia particulata (PM) durante a diffusione di l'infezzione,[5,6] in modu coerente cù l'evidenza digià
dispunibule per altri virus.[7-15] Tuttavia, u prublema di u microbioma assuciatu à u PM in l'aria, in particulare in l'ambienti urbani, ferma largamente investigatu, [16] è - à l'oghje - nimu hà ancu realizatu studii sperimentali specificamente destinati. à cunfirmà o escludendu a prisenza di u SARS-CoV-2 in PM.
Quì, prisentamu i primi risultati di l'analisi chì avemu realizatu nantu à 34 campioni PM10 di PM10 outdoor/airborne da un situ industriale di a Pruvincia di Bergamo, raccolti cù dui campioni d'aria diffirenti per un periudu continuu di 3 settimane, da u 21 di ferraghju à marzu. 13.
Dopu a metodulugia descritta da Pan et al.in u 2019 (per a cullizzioni, a dimensione di e particelle è a rilevazione di virus in l'aria), [17] campioni di PM sò stati raccolti nantu à filtri di fibra di quartz cù un sampler gravimetricu di l'aria di pocu voluminu (38,3 l/min per 23 h), in cunfurmità cù u metudu di riferimentu EN12341. : 2014 per u monitoraghju PM10.A materia particulata hè stata intrappulata nantu à i filtri cù u 99,9% tipicuritenzione di aerosol, cunservatu currettamente è consegnatu à u laboratoriu di Genomica Applicata è Comparativa di l'Università di Trieste.Data a natura "ambientale" di a mostra, presumibbilmente ricca in inhibitori di DNA polimerasi, avemu procedutu à l'estrazione di RNA utilizendu u kit di microbi di terra fecale Quick RNA adattatu à u tipu di filtri.[18]A mità di filtru hè stata rotolata, cù a parte superiore rivolta versu l'internu,in un tubu di polipropilene da 5 ml, inseme cù e perle furnite in u kit.Da l'iniziali 1 ml di lysisbuffer, avemu pussutu ottene circa 400 ul di suluzione, chì hè stata poi processata cum'è definitu da i protokolli standard, risultatu in un eluate finali di 15 ul.In seguitu, 5 ul sò stati utilizati per a prova SARS-CoV-2.Data l'origine particulari di a mostra, u qScript XLT 1-Step RT-qPCR ToughMix hè stata utilizata.[19]I sistemi di amplificazione eranu quelli di u protocolu sviluppatu da Corman et al, publicatu in u situ web di l'OMS [20].
A prova hè stata esplicitamente destinata à cunfirmà o esclude a prisenza di u SARS-CoV-2 RNA nantu à a materia particulata.U primu analisi utilizatu u "genu E" cum'è un marcatu molekulari è pruducia un risultatu pusitivu impressiunanti nantu à 15 di 16 filtri ancu s'è, cum'è pudemu aspittà, u Ct era trà 36-38 ciculi.
Dopu questu, avemu replicatu l'analisi nantu à 6 di i filtri pusitivi (dighjà pusitivi à u "genu E") utilizendu u "genu RtDR" cum'è un marcatore moleculare - chì hè assai specificu per SARS-CoV-2 - ghjunghje à 5 risultati significativi. di pusitivi;i testi di cuntrollu per escludiri a falsa pusitivita sò ancu realizati cù successu (Fig. 1).
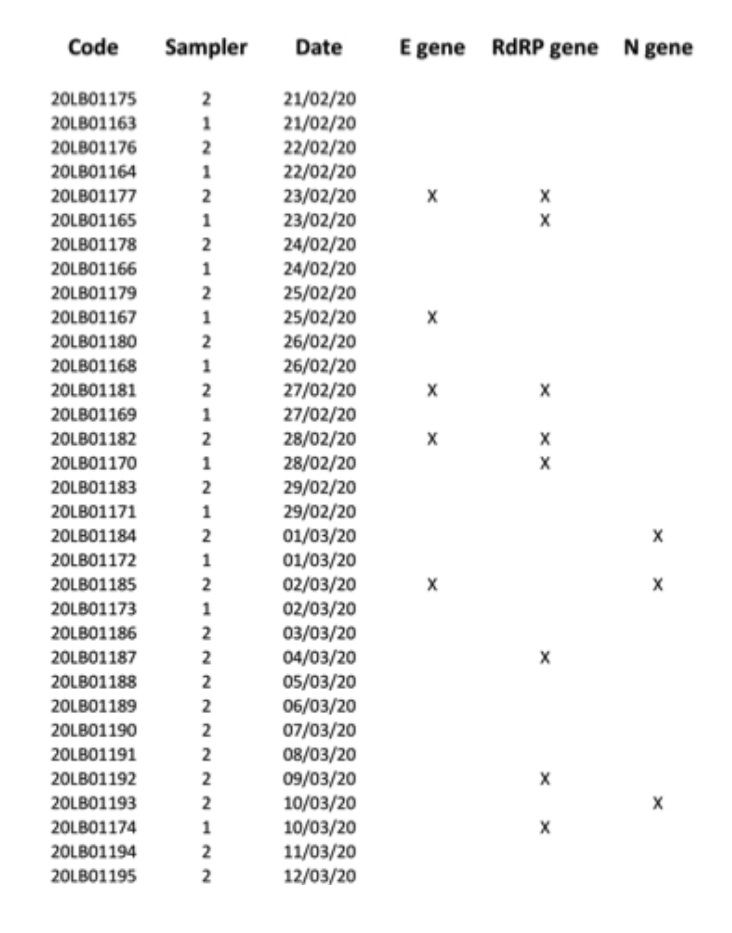
Per evità l'esaurimentu di u scarsu materiale di campionamentu dispunibule, i restanti RNA estratti sò stati consegnati à l'Hospital Universitariu lucale (unu di i centri clinichi autorizati da u Guvernu talianu per i testi diagnostichi SARS-CoV-2), per fà una seconda prova a cieca parallela.Stu secondu laboratoriu clinicu hà pruvatu l'estrazzioni di RNA 34 per i geni E, N è RdRP, rapprisentanu 7 risultati pusitivi per almenu unu di i trè geni di marcatori, cù positività cunfirmata separatamente per tutti i trè marcatori (Fig. 2).A causa di a natura di a mostra, è cunsiderendu chì u campionamentu ùn hè statu realizatu per scopi diagnostichi clinichi, ma per teste di contaminazione ambientale (piendu ancu in contu chì i filtri sò stati conservati per almenu quattru settimane prima di esse sottumessi à l'analisi genetica moleculare, cum'èuna cunsequenza di u chjusu talianu), pudemu cunfirmà avè dimustratu raghjone a prisenza di l'RNA virali SARS-CoV-2 detectendu un "genu RtDR" altamente specificu nantu à 8 filtri.Tuttavia, per via di a mancanza di materiali supplementarii da i filtri, ùn avemu micca pussutu ripetiri un numeru abbastanza di teste per dimustrà positività per tutti i marcatori moleculari 3 simultaneamente.
Questa hè a prima evidenza preliminare chì l'ARN SARS-CoV-2 pò esse presente nantu à a materia particulata esterna, suggerendu cusì chì, in cundizioni di stabilità atmosferica è concentrazioni elevate di PM, SARS-CoV-2 puderia creà clusters cù PM all'aperto è - da riducendu u so coefficient di diffusione - rinfurzà a persistenza di u virus in l'atmosfera.Ulteriori cunfirmazioni di stu preliminariuL'evidenza hè in corso, è deve include una valutazione in tempu reale nantu à a vitalità di u SARS-CoV-2 è a so virulenza quandu hè adsorbitu nantu à a materia particulata.Attualmente, ùn si pò fà alcuna ipotesi in quantu à a correlazione trà a presenza di u virus nantu à a progressione di u focu PM è COVID-19.L'altri prublemi da esse specificamente affrontati sò a concentrazione media di PM eventualmentenecessaria per un potenziale "effettu di spinta" di u contagionu (in casu hè cunfirmatu chì PM puderia agisce cum'è "portatore" per i nuclei di gocce virali), o ancu a pussibilità teorica di immunizazione cunsequente à esposizioni di dosi minime à soglie più basse di PM. .
Fig.1 Curves d'amplificazione di i geni E (A) è RdRP (B): e linee verdi rapprisentanu i filtri testati;linee incruciaterapprisenta l'estrazzioni di filtri di riferimentu;e linee rosse rapprisentanu l'amplificazione di i campioni pusitivi.
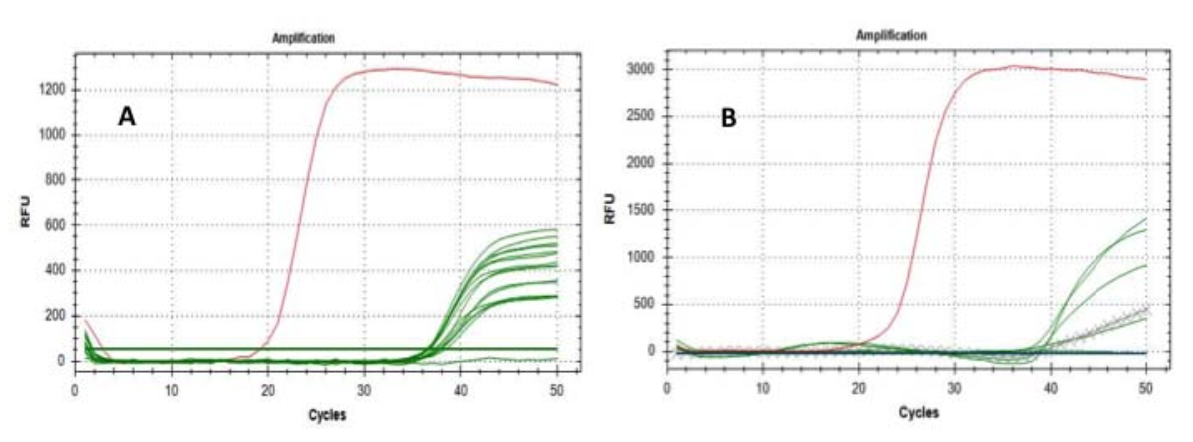
Fig.2.Risultati pusitivi (marcati cù X) per i geni E, N è RdRP ottenuti per tutti i campioni di 34 PM10i filtri testati in a seconda analisi parallela.
 Leonardo Setti1, Fabrizio Passarini2, Gianluigi De Gennaro3, Pierluigi Barbieri4, Maria Grazia Perrone5, Massimo Borelli6, Jolanda Palmisani3, Alessia Di Gilio3, Valentina Torboli6, Alberto Pallavicini6, Maurizio Ruscio7, Prisco Piscitelli8, Alessandro Miani8,9
Leonardo Setti1, Fabrizio Passarini2, Gianluigi De Gennaro3, Pierluigi Barbieri4, Maria Grazia Perrone5, Massimo Borelli6, Jolanda Palmisani3, Alessia Di Gilio3, Valentina Torboli6, Alberto Pallavicini6, Maurizio Ruscio7, Prisco Piscitelli8, Alessandro Miani8,9
1. Dipartimentu di Chimica Industriale, Università di Bologne, Viale del Risorgimento – 4, I-40136, Bologne, Italie
e-mail: leonardo.setti@unibo.it
2. Centru Interdipartimentale per a Ricerca Industriale "Fonti Rinnovabili, Ambiente, Crescita Blu, Energia",
University of Bologna, Rimini, Italy e-mail: fabrizio.passarini@unibo.it
3. Dipartimentu di Biologia, Università “Aldo Moro” di Bari, Bari, Italia
e-mail: gianluigi.degennaro@uniba.it; alessia.digilio@uniba.it; jolanda.palmisani@uniba.it
4. Dipartimentu di Scienze Chimiche è Farmaceutiche, Università di Trieste, Trieste, Italia
e-mail: barbierp@units.it
5. Divisione di Ricerca Ambientale, TCR TECORA, Milanu, Italia
e-mail: mariagrazia.perrone@tcrtecora.com
6. Dipartimentu di Scienze di a Vita – Università di Trieste, Trieste, Italia
e-mail: borelli@units.it; torboli@units.it; pallavic@units.it
7. Divisione di Medicina di Laboratoriu, Hospital Universitario Giuliano Isontina (ASU GI), Trieste, Italia
email: maurizio.ruscio@asugi.sanita.fvg.it
8. Società Italiana di Medicina Ambientale (SIMA), Milan, Italia
e-mail: priscofreedom@hotmail.com; alessandro.miani@unimi.it
9. Dipartimentu di Scienza Ambientale è Poicy, Università di Milano, Milanu, Italia
e-mail: priscofreedom@hotmail.com; alessandro.miani@unimi.it
Auteur Correspondant :
Leonardo Setti, Department of Industrial Chemistry, University of Bologna Viale del Risorgimento 4, 40136, Bologna, Italy; e-mail: leonardo.setti@unibo.it
Referenze
1. Organizazione Mondiale di a Salute, Modi di trasmissione di virus chì causanu COVID-19: implicazioni per i cunsiglii di precauzione IPC, Breve scientificu;dispunibule à: https://www.who.int/newsroom/commentaries/detail/modes-of-transmission-of-virus-causing-covid-19-implications-for-ipcprecaution-recommendations (29 di marzu 2020)
2. Ministeru di a Salute talianu, bollettinu quotidianu epidemia di Covid-19 in Italia, dispunibule à http://www.salute.gov.it/imgs/C_17_notizie_4451_0_file.pdf
3. EEA, Agenzia Ambientale Europea, Rapportu 2019 Air Quality in Europe;nru 10/2019;Agenzia Europea di l'Ambiente: Copenhagen, Danimarca, dispunibule à: https://www.eea.europa.eu/publications/airquality-in-europe-2019
4. Xiao Wu, Rachel C. Nethery, M. Benjamin Sabath, Danielle Braun, Francesca Dominici, Exposure to air pollution and COVID-19 mortality in the United States, dispunibule à: https://projects.iq.harvard.edu/ files/covid-pm/files/pm_and_covid_mortality.pdf
5. Società Italiana di Medicina Ambientale (SIMA), Position Paper Particulate Matter è COVID-19,
dispunibule à: http://www.simaonlus.it/wpsima/wp-content/uploads/2020/03/COVID_19_positionpaper_ENG.pdf
6. Setti L., Passarini F., De Gennaro G., Barbieri P., Perrone MG, Piazzalunga A., Borelli M., Palmisani J., Di Gilio A, Piscitelli P, Miani A., Is there a Plausible Role per Particulate Matter in a diffusione di COVID-19 in l'Italia di u Nordu ?, BMJ Rapid Responses, 8 d'aprile 2020, dispunibule à: https://www.bmj.com/content/368/bmj.m1103/rapid-responses
7. Sedlmaier, N., Hoppenheidt, K., Krist, H., Lehmann, S., Lang, H., Buttner, M. Generation of avian influenza virus (AIV) contaminated fecal fine particulate matter (PM2.5): rilevazione di genoma è infettività è calculu di immissione.Microbiologia Veterinaria.139, 156-164 (2009)
8. Zhao, Y., Richardson, B., Takle, E., Chai, L., Schmitt, D., Win, H. A trasmissione Airborne pò avè avutu un rolu in a diffusione di l'epidemie di influenza aviaria altamente patogena 2015 in u Stati Uniti.Sci Rep. 9, 11755. https://doi.org/10.1038/s41598-019-47788-z (2019)
9. Ma, Y., Zhou, J., Yang, S., Zhao, Y., Zheng, X. Assessment for the impact of dust events on saramples incidence in Western China.Ambiente atmosfericu.157, 1-9 (2017)
10. Sorensen, JH, Mackay, DKJ, Jensen, C. Ø., Donaldson, AI Un mudellu integratu per predice a diffusione atmosferica di u virus epidemiol.Infect., 124, 577–590 (2000)
11. Glostera, J., Alexandersen, S. New Directions: Airborne Transmission of Foot-and-Mouth Disease Virus Atmospheric Environment, 38 (3), 503-505 (2004)
12. Reche, I., D'Orta, G., Mladenov, N., Winget, DM, Suttle, CA Tassi di Depositu di virus è battìri sopra à a capa di frontiera atmosferica.U Journal ISME.12, 1154-1162 (2018)
13. Qin, N., Liang, P., Wu, C., Wang, G., Xu, Q., Xiong, X., Wang, T., Zolfo, M., Segata, N., Qin, H. ., Knight, R., Gilbert, JA, Zhu, TF Indagine longitudinale di u microbioma assuciatu à a materia particulata in una megacity.Biologia di u genoma.21, 55 (2020)
14. Zhao, Y., Richardson, B., Takle, E., Chai, L., Schmitt, D., Win, H. A trasmissione Airborne pò avè
hà ghjucatu un rolu in a diffusione di l'epidemie di influenza aviaria altamente patogena di u 2015 in i Stati Uniti.Sci
Rep. 9, 11755. https://doi.org/10.1038/s41598-019-47788-z (2019)
15. Ma, Y., Zhou, J., Yang, S., Zhao, Y., Zheng, X. Assessment for the impact of dust events on saramples incidence in Western China.Ambiente atmosfericu.157, 1-9 (2017)
16. Jiang, W., Laing, P., Wang, B., Fang, J., Lang, J., Tian, G., Jiang, J., Zhu, TF Estrazione di DNA ottimizzata è sequenza metagenomica di e cumunità microbiche di l'aria. .Nat.Protoc.10, 768-779 (2015)
17. Pan, M., Lednicky, JA, Wu, C.-Y., Collection, particle sizing and detection of airborne viruses.Journal of Applied Microbiology, 127, 1596-1611 (2019)
18. Zymoresearch Ldt, descrizzione di u produttu, dispunibule à: https://www.zymoresearch.com/products/quick-rnafecal-soil-microbe microprep-kit
19. Quantabio Ltd, descrizzione di u pruduttu, dispunibule à: https://www.quantabio.com/qscript-xlt-1-steprt-qpcr-toughmix
20. Corman, VM, Landt, O., Kaiser, M., Molenkamp, R., Meijer, A., Chu, DK, & Mulders, DG (2020).
Rilevazione di u novu coronavirus 2019 (2019-nCoV) da RT-PCR in tempu reale.Eurosurveillance, 25(3), dispunibule à:.https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6988269/
Originale: https://doi.org/10.1101/2020.04.15.20065995
Tempu di post: Apr-18-2020
